Questo report descrive le caratteristiche genetiche di ulteriori 98 campioni di SARS-CoV-2 (6 storici e 92 sorveglianza) identificati in Veneto, per un totale di 154 campioni prelevati tra novembre 2020 e gennaio 2021 (sorveglianza) e 6 campioni raccolti tra giugno e ottobre 2020 (storici).
Su mandato regionale l’Istituto Zooprofilattico Sperimentale delle Venezie (IZSVe) sta monitorando le caratteristiche genetiche e la variabilità dei ceppi di SARS-CoV-2 presenti in Veneto. L’emergere di mutazioni nel genoma di agenti virali ad RNA come SARS-CoV-2 è un evento naturale ed atteso. Cambiamenti nella trasmissibilità del virus, nella gravità della malattia, nella capacità del virus di sfuggire all’immunità acquisita (post-infezione o vaccinazione) e ai test diagnostici in uso: questi sono gli elementi cruciali che definiscono le dinamiche di interazione di SARS-CoV-2 con la popolazione ospite. Sequenziare il genoma di un virus significa poter riconoscere l’emergere di varianti virali che possono modificare l’andamento e l’impatto dell’epidemia. Le mutazioni più interessanti sono a livello della proteina Spike del virus data l’importanza che questa riveste per il legame con i recettori cellulari e perché verso di essa sono rivolti i principali anticorpi che danno la protezione verso l’infezione e le forme cliniche.
Descrizione delle varianti identificate
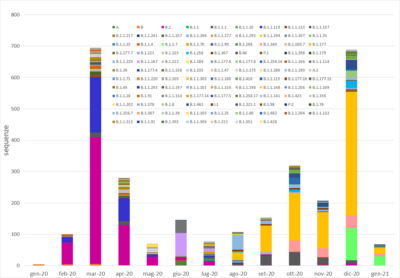
SARS-CoV-2 è stato classificato in lineage. Ciascun lineage contiene un gruppo di genomi virali caratterizzati da uno specifico set di mutazioni. I virus caratterizzati in Veneto da novembre 2020 appartengono a quindici diversi lineage (Rambaut et al., 2020): B.1, B.1.1.1, B.1.1.10, B.1.1.217, B.1.1.241, B.1.1.307, B.1.160, B.1.160.7, B.1.177, B.1.177.7, B.1.221, B.1.258, B.1.367, B.1.1.7 e A (Figura 1), di cui cinque (sottolineati) appartengono a varianti selezionate dal Centro Europeo per la prevenzione e controllo delle malattie (ECDC) come varianti che destano preoccupazione e da monitorare con attenzione, o più precisamente “variants of current concern”.
La variante inglese (lineage B.1.1.7 o VOC-202012/01) è stata identificata in 17 campioni (per 6 è stato sequenziato solo il gene della Spike e per 11 è stato ottenuto il genoma completo). Nessuno dei campioni analizzati finora appartiene alla variante sudafricana (501.V2) o a quella brasiliana (P.1).
Nelle figure 2-4 è mostrata la distribuzione dei vari lineage in Italia o in Veneto.
Di seguito la descrizione dei lineage identificati in Veneto e le mutazioni identificate nella proteina Spike.
- Lineage A (N=6 sequenze)
Si tratta di un lineage circolante in Europa sin dall’inizio della pandemia. In Italia è stato identificato solo in Sicilia e in Veneto nella provincia di Treviso (novembre-gennaio).
Mutazioni da segnalare nella proteina Spike: D614N. - Lineage B.1.1.7 o VOC-202012/01 o 20I/501Y.V1 (N=11 sequenze, genoma completo)
Lineage a cui appartengono i virus della variante inglese VOC-202012/01 o 20I/501Y.V1 identificata nel Regno Unito a partire da settembre. Questo lineage è stato identificato per la prima volta nel territorio italiano a dicembre. Si tratta di una variante di particolare interesse in quanto sembra essere caratterizzata da una maggiore trasmissibilità. Tale variante si contraddistingue per 17 mutazioni (14 sostituzioni amminoacidiche e 3 delezioni) lungo il genoma.
Mutazioni caratterizzanti nella proteina Spike: delezione 69-70, delezione 144, N501Y, A570D, P681H, T716I, S982A, D1118H.
Altre mutazioni aggiuntive nella proteina Spike individuate nei campioni veneti: A222V presente in 1/11 sequenze, L5F presente in 1/11 sequenze e N710V presente in 1/11 sequenze. - Lineage B.1.1.1 (N=8 sequenze)
Si tratta di un lineage diffuso nel territorio Italiano, identificato a partire da marzo 2020.
Mutazioni da segnalare nella proteina Spike: D614G. - Lineage B.1.1.217 (N=1 sequenza)
Identificato in Italia (Veneto) per la prima volta ad agosto secondo i dati disponibili in GISAID in data 29/01/2021. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: D614G. - Lineage B.1.1.241 (N=2 sequenze)
Identificato in Italia (Campania) per la prima volta ad agosto, secondo i dati disponibili in GISAID in data 29/01/2021. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: A352S (presente in 1/2 sequenze), D614G. - Lineage B.1.160 e B.1.160.7 o 20A.EU2 (N=28 sequenze)
Identificato nel territorio italiano a partire da aprile, secondo i dati disponibili in GISAID in data 29/01/2021. Virus appartenenti a questo lineage sono stati identificati da marzo in diversi paesi europei, in particolare Regno Unito, Francia e Svizzera. Il lineage B.1.160 ha visto un rapido incremento in Francia all’inizio della seconda ondata (probabilmente come conseguenza del cosiddetto founder effect).
Mutazioni da segnalare nella proteina Spike: S477N, D614G.
- Lineage B.1.177 e B.1.177.7 o 20A.EU1 (N=81 sequenze)
Identificato nel territorio italiano a partire da giugno. Virus appartenenti a questo lineage sono stati identificati da marzo in diversi paesi europei, in particolare Regno Unito, Spagna e Danimarca. Il lineage B.1.177 ha visto un rapido incremento in Spagna all’inizio della seconda ondata e successivamente nel resto d’Europa (Hodcroft et al., 2020).
Mutazioni da segnalare nella proteina Spike: A222V, D614G, A262S+P272L (presenti in 23/81 sequenze), K356E (presente in 1/81 sequenze) - Lineage B.1.221 (N=6 sequenze)
Identificato nel territorio italiano a partire da agosto. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: D614G. - Lineage B.1.258 (N=5 sequenze)
Identificato nel territorio italiano a partire da agosto. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: delezione 69-70 (in 3/5 virus), N439K, D614G. - Lineage B.1 (N=2 sequenze)
Identificato nel territorio italiano a partire da febbraio 2020. È il lineage che ha circolato con la più alta frequenza in Italia e in Veneto durante la prima onda epidemica.
Mutazioni da segnalare nella proteina Spike: D614G, E516Q (in 1/2 sequenze). - Lineage B.1.1.10 (N=1 sequenza)
Identificato per la prima volta nel territorio italiano a gennaio 2021 (dato riportato per la prima volta con questo report) in un paziente di rientro dal Libano. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei, in particolare nel corso della prima ondata epidemica.
Mutazioni da segnalare nella proteina Spike: L452R, D614G. - Lineage B.1.1.307 (N=1 sequenza)
Identificato per la prima volta nel territorio italiano a dicembre 2020 (dato riportato per la prima volta con questo report). Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei, in particolare nel Regno Unito dalla seconda metà del 2020.
Mutazioni da segnalare nella proteina Spike: D614G. - Lineage B.1.367 (N=2 sequenze)
Identificato nel territorio italiano (Veneto incluso) a partire da agosto 2020. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei, in particolare in Francia e Regno Unito dalla seconda metà del 2020.
Mutazioni da segnalare nella proteina Spike: D614G.
Effetto delle mutazioni più importanti nella proteina Spike
- A222V: caratteristica del lineage1.177, il quale mostra un notevole aumento di frequenza a livello nazionale e regionale. Non ci sono dati scientifici che chiariscono l’effetto di questa mutazione sulle caratteristiche biologiche del virus.
- A352S: mutazione nel punto di attacco della proteina S al recettore cellulare o dominio di legame al recettore.
- K356E: mutazione nel dominio di legame al recettore. Identificata in un campione di novembre del lineage1.177. L’amminoacido in posizione 356 della Spike cade all’interno di un sito potenzialmente riconosciuto da parte di anticorpi neutralizzanti (Huang et al., 2020). Si tratta di una mutazione raramente osservata nelle sequenze di SARS-CoV-2 e il suo effetto non è mai stato descritto.
- N439K: mutazione nel dominio di legame al recettore. Identificata nei campioni appartenenti al lineage1.258. è stata associata ad un aumento dell’affinità per il recettore umano ACE2 e alla capacità di sfuggire all’azione di alcuni anticorpi monoclonali e di ridurre l’attività neutralizzante del siero di una parte di individui guariti dall’infezione (Thomson et al., 2021; Li et al., 2020; Hodcroft et al., 2020; SSI, 2020).
- L452R: mutazione nel dominio di legame al recettore. Identificata in un campione appartenente al lineage B.1.1.10 prelevato da un paziente di rientro dal Libano. Questa mutazione è stata associata alla capacità di sfuggire all’azione di alcuni anticorpi monoclonali (Li et al., 2020; Wang et al., 2020). In particolare, la posizione 452 è uno dei siti di legame dell’anticorpo monoloclonale LY3819253/LY-CoV555 o bamlanivimab (Jones et al., preprint). A gennaio 2021 è stato osservato in California un notevole aumento della frequenza di tale mutazione associata a virus responsabili di numerosi cluster di infezione.
- S477N: mutazione nel dominio di legame al recettore. Identificata in virus appartenenti al lineage1.160 e B.1.160.7. Questa mutazione sembra essere associata ad un aumento dell’affinità per il recettore umano ACE2 (Grid Based Pharmacophore Model) e alla possibilità di eludere l’attività neutralizzante di alcuni anticorpi monoclonali (Ortuso et al., 2020; Singh et al., 2020; Liu et al., 2020).
- N501Y: mutazione nel dominio di legame al recettore. Identificata in tutti i campioni appartenenti al lineage B.1.1.7. è stata associata ad un aumento dell’affinità per il recettore umano ACE2 (Starr et al., 2020).
- E516Q: mutazione nel dominio di legame al recettore. Identificata in un campione appartenente al lineage1. Tale mutazione cade all’interno di un epitopo riconosciuto da parte di anticorpi monoclonali (Wu et al., 2020).
- D614G: si tratta di una mutazione diffusa fin dalla prima ondata. è stata associata ad un aumento della trasmissibilità del virus (Volz et al., 2020).
- D614N: il ruolo di questa mutazione non è ancora chiaro, ma similmente alla D614G, sembra aumentare la stabilità del trimero della proteina Spike (Juraszek et al. et al., 2021).
Figure

Figura 3. Distribuzione mensile dei principali lineage circolanti. In alto il dato relativo all’Italia mentre in basso quello relativo alla sola regione Veneto. Si può notare in entrambi i grafici che i genotipi B.1 e B.1.1.119 non sono più stati rilevati nella seconda metà del 2020 mentre sono diventati prevalenti i genotipi B.1.177 e B.1.160 (dati basati sulle sequenze italiane disponibili in GISAID in data 29/01/2021).
Referenze
- Wu Y, Li C, Xia S, Tian X, Kong Y, Wang Z, Gu C, Zhang R, Tu C, Xie Y, Yang Z, Lu L, Jiang S, Ying T. Identification of Human Single-Domain Antibodies against SARS-CoV-2. Cell Host Microbe. 2020 Jun 10;27(6):891-898.e5. doi: 10.1016/j.chom.2020.04.023. Epub 2020 May 14. PMID: 32413276; PMCID: PMC7224157.
- Jones BE, Brown-Augsburger PL, Corbett KS, Westendorf K, Davies J, Cujec TP, Wiethoff CM, Blackbourne JL, Heinz BA, Foster D, Higgs RE, Balasubramaniam D, Wang L, Bidshahri R, Kraft L, Hwang Y, Žentelis S, Jepson KR, Goya R, Smith MA, Collins DW, Hinshaw SJ, Tycho SA, Pellacani D, Xiang P, Muthuraman K, Sobhanifar S, Piper MH, Triana FJ, Hendle J, Pustilnik A, Adams AC, Berens SJ, Baric RS, Martinez DR, Cross RW, Geisbert TW, Borisevich V, Abiona O, Belli HM, de Vries M, Mohamed A, Dittmann M, Samanovic M, Mulligan MJ, Goldsmith JA, Hsieh CL, Johnson NV, Wrapp D, McLellan JS, Barnhart BC, Graham BS, Mascola JR, Hansen CL, Falconer E. LY-CoV555, a rapidly isolated potent neutralizing antibody, provides protection in a non-human primate model of SARS-CoV-2 infection. bioRxiv [Preprint]. 2020 Oct 1:2020.09.30.318972. doi: 10.1101/2020.09.30.318972. PMID: 33024963; PMCID: PMC7536866.
- Wang, L., Wang, L. & Zhuang, H. Profiling and characterization of SARS-CoV-2 mutants’ infectivity and antigenicity. Sig Transduct Target Ther 5, 185 (2020). https://doi.org/10.1038/s41392-020-00302-8
- Yang Huang, Hui Sun, Hai Yu, Shaowei Li, Qingbing Zheng, Ningshao Xia, Neutralizing antibodies against SARS-CoV-2: current understanding, challenge and perspective, Antibody Therapeutics, Volume 3, Issue 4, December 2020, Pages 285–299, https://doi.org/10.1093/abt/tbaa028
- Bal A, Destras G, Gaymard A, Regue H, Semanas Q, d’Aubarde C, Billaud G, Laurent F, Gonzales C, Valette M, Lina B, Morfin F, Josset L. Screening of the H69 and V70 deletions in the SARS-CoV-2 spike protein with a RT-PCR diagnosis assay reveals low prevalence in Lyon, France. medRxiv 2020, 2020.11.10.20228528.
- Kemp, Steven A., Dami A. Collier, Rawlings Datir, Salma Gayed, Aminu Jahun, Myra Hosmillo, Isabella Atm Ferreira, et al. 2020. Neutralising Antibodies Drive Spike Mediated SARS-CoV-2 Evasion. medRxiv. https://www.medrxiv.org/content/10.1101/2020.12.05.20241927v1.full.
- Andrew Rambaut, Edward C. Holmes, Áine O’Toole, Verity Hill, John T. McCrone, Christopher Ruis, Louis du Plessis, and Oliver G. Pybus. 2020. A Dynamic Nomenclature Proposal for SARS-CoV-2 Lineages to Assist Genomic Epidemiology. Nature Microbiology 5 (11): 1403–7.
- Thomson, E. C., Rosen, L. E., Shepherd, J. G., Spreafico, R., da Silva Filipe, A., Wojcechowskyj, J. A., Davis, C., Piccoli, L., Pascall, D. J., Dillen, J., Lytras, S., Czudnochowski, N., Shah, R., Meury, M., Jesudason, N., De Marco, A., Li, K., Bassi, J., O’Toole, A., Pinto, D., … Snell, G. (2021). Circulating SARS-CoV-2 spike N439K variants maintain fitness while evading antibody-mediated immunity. Cell, Advance online publication. https://doi.org/10.1016/j.cell.2021.01.037
- Zhuoming Liu, Laura A. VanBlargan, Paul W. Rothlauf, Louis-Marie Bloyet, Rita E. Chen, Spencer Stumpf, Haiyan Zhao, John M. Errico, Elitza S. Theel, Ali H. Ellebedy, Daved H. Fremont, Michael S. Diamond, Sean P. J. Whelan. Landscape analysis of escape variants identifies SARS-CoV-2 spike mutations that attenuate monoclonal and serum antibody neutralization. bioRxiv 2020.11.06.372037
- Francesco Ortuso, Daniele Mercatelli, Pietro Hiram Guzzi, Federico Manuel Giorgi. Structural Genetics of circulating variants affecting the SARS-CoV-2 Spike / human ACE2 complex. bioRxiv 2020.09.09.289074; doi: https://doi.org/10.1101/2020.09.09.289074
- Starr TN, Greaney AJ, Hilton SK, Ellis D, Crawford KHD, Dingens AS, Navarro MJ, Bowen JE, Tortorici MA, Walls AC, King NP, Veesler D, Bloom JD. Deep Mutational Scanning of SARS-CoV-2 Receptor Binding Domain Reveals Constraints on Folding and ACE2 Binding. Cell. 2020 Sep 3;182(5):1295-1310.e20. doi: 10.1016/j.cell.2020.08.012.
- Erik Volz, Verity Hill, John T. McCrone, Anna Price, David Jorgensen, Áine O’Toole, Joel Southgate, et al. 2020. Evaluating the Effects of SARS-CoV-2 Spike Mutation D614G on Transmissibility and Pathogenicity. Cell, November. https://doi.org/10.1016/j.cell.2020.11.020.
- Juraszek J, Rutten L, Blokland S, Bouchier P, Voorzaat R, Ritschel T, J. G. Bakkers M, L. R. Renault L & P. M. Langedijk J. Stabilizing the closed SARS-CoV-2 spike trimer. Nature Communications (2021). https://www.nature.com/articles/s41467-020-20321-x#MOESM1
- Statens Serum Institut (SSI). Beskrivelse af covid-19-variant N439K.22 December 2020. Available from: https://www.ssi.dk/aktuelt/nyheder/2020/beskrivelse-af-covid-19-variant-n439k
- Hodcroft EB, Zuber M, Nadeau S, Crawford KHD, Bloom JD, Veesler D, et al. Emergence and spread of a SARS-CoV-2 variant through Europe in the summer of 2020. medRxiv [Preprint]. 27 November 2020; Available from: https://www.medrxiv.org/content/10.1101/2020.10.25.20219063v2
- Li Q, Wu J, Nie J, Zhang L, Hao H, Liu S, et al. The Impact of Mutations in SARS-CoV-2 Spike on Viral Infectivity and Antigenicity. Cell. 2020;182(5):1284-94. Available from: https://www.sciencedirect.com/science/article/pii/S0092867420308771
- Starr TN, Greaney AJ, Hilton SK, Ellis D, Crawford KHD, Dingens AS, et al. Deep Mutational Scanning of SARS-CoV-2 Receptor Binding Domain Reveals Constraints on Folding and ACE2 Binding. Cell. 2020;182(5):1295-310. Available from: https://www.sciencedirect.com/science/article/pii/S0092867420310035
- Amit Singh, Georg Steinkellner, Katharina Köchl, Karl Gruber, Christian C. Gruber. Serine 477 plays a crucial role in the interaction of the SARS-CoV-2 spike protein with the human receptor ACE2. November 2020. Preprint from Researchsquare; doi: 10.21203/rs.3.rs-106969/v2.