Su mandato regionale l’Istituto Zooprofilattico Sperimentale delle Venezie (IZSVe) sta monitorando le caratteristiche genetiche e la variabilità dei ceppi di SARS-CoV-2 presenti in Veneto. Nelle ultime settimane è stata effettuata la sequenza del genoma completo di 37 ceppi SARS-CoV-2 identificati nelle province di Verona, Vicenza e Rovigo tra il 2 novembre e il 4 dicembre.
Questi virus appartengono a otto diversi lineage: B.1.1.1, B.1.1.217, B.1.1.241, B.1.160, B.1.160.7, B.1.177, B.1.221, B.1.258. Il 79% dei campioni appartengono ai lineage B.1.177 (60%) e B.1.160 (19%). Sulla base delle sequenze disponibili in GISAID in data 23 dicembre 2020, questi due lineage rappresentano attualmente le varianti più diffuse sia nella regione Veneto sia a livello nazionale (figure 1 e 2).
In data 24 dicembre è stata inoltre riscontrata la presenza di virus potenzialmente ascrivibili alla “variante inglese” VUI 202012/01 (B1.1.7 lineage) in campioni positivi SARS-CoV-2, prelevati in provincia di Treviso e Vicenza da pazienti provenienti dall’Inghilterra, dove tale variante sembra si sia originata e diffusa molto velocemente. La presenza di tale variante nei campioni esaminati è stata ipotizzata grazie all’identificazione di specifiche mutazioni sul gene S (che codifica per la proteina Spike) caratterizzanti la VUI 202012/01.
Tutti i dati genetici del virus finora raccolti costituiscono una fotografia, seppur parziale, delle varianti rilevate in Veneto nel periodo sopra indicato. Si sottolinea che un’interpretazione completa sulla dinamica di diffusione del SAR-CoV-2 deve essere opportunamente contestualizzata anche alla luce dei dati epidemiologici rilevati sul territorio e delle variabili che condizionano la diffusione dei virus in una determinata area geografica.
Si ringraziano tutti i laboratori della Regione che hanno fornito e continuano a fornire i campioni necessari allo studio dei SARS-CoV-2 circolanti in Veneto.
Descrizione delle varianti indentificate
Analisi filogenetica (Pangolin lineage)
I virus analizzati appartengono a otto diversi Pangolin lineage (B.1.1.1, B.1.1.217, B.1.1.241, B.1.160, B.1.160.7, B.1.177, B.1.221, B.1.258). Il 79% dei campioni appartengono ai lineage B.1.177 (60%) e B.1.160 (19%). Questi lineage mostrano un notevole aumento di frequenza sia a livello nazionale che nella regione Veneto nel mese di Novembre (figura 2).
Nessuno dei 37 campioni analizzati prima del 24 Dicembre 2020 appartiene al lineage B.1.1.7 (variante VUI-202012/01 identificata nel Regno Unito) e al lineage B.1.351 (variante identificata in Sud Africa).
- Lineage B.1.1.1 (N=1)
Si tratta di uno dei lineage più diffusi nel territorio Italiano identificato in Veneto da Marzo 2020.
Mutazioni da segnalare nella proteina Spike: D614G - Lineage B.1.1.217 (N=1)
Non ancora descritto in Italia secondo i dati disponibili in GISAID in data 23/12/2020. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: D614G - Lineage B.1.1.241 (N=1)
Non ancora descritto in Italia secondo i dati disponibili in GISAID in data 23/12/2020. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: A352S, D614G - Lineage B.1.1.160 e B.1.1.160.7 (N=8)
Identificato nel territorio italiano a partire da novembre. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: S477N, D614G
- Lineage B.1.1.177 (N=22)
Identificato nel territorio italiano a partire da agosto. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: A222V, D614G - Lineage B.1.1.221 (N=3)
Identificato nel territorio italiano a partire da novembre. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: D614G - Lineage B.1.1.258 (N=1)
Identificato nel territorio italiano a partire da ottobre. Virus appartenenti a questo lineage sono stati identificati in precedenza in diversi paesi europei.
Mutazioni da segnalare nella proteina Spike: delezione 69-70, N439K, D614G
Analisi delle mutazioni
Abbiamo identificato un totale di 135 siti variabili lungo il genoma dei campioni analizzati rispetto al ceppo Wuhan-hu-1: 131 mutazioni amminoacidiche e quattro delezioni in ORF 7b, ORF 3a, ORF 8 e Spike. Il 95% di queste mutazioni sono state precedentemente identificate in altri SARS-CoV-2, mentre 7 sono uniche dei campioni analizzati.
A livello della proteina Spike sono state identificate 23 mutazioni, tre di queste a livello del sito recettoriale. In particolare segnaliamo:
- Delezione 69-70. Identificata in un singolo campione appartenente al lineage B.1.258. Questa delezione potrebbe essere associata all’evasione della risposta immunitaria dell’ospite e potrebbe compromettere il risultato di alcuni test diagnostici che hanno come target il gene S, come il saggio TaqPath a tre target (ThermoFisher) e BioFire a due target (Biomerieux).
- A222V. Identificata nei 22 campioni del lineage B.1.177, il quale mostra un notevole aumento di frequenza a livello nazionale e regionale. Tale lineage sembra essersi originato in Spagna, da dove si è diffuso nel resto dell’Europa. Tuttavia non ci sono chiare evidenze sull’effetto di questa mutazione. 7/22 campioni appartenenti al lineage B.1.177 presentano anche le mutazioni A262S e P272L.
- A352S. Mutazione nel dominio di legame al recettore. Identificata in un singolo campione appartenente al lineage B.1.241.
- N439K. Mutazione nel dominio di legame al recettore. Identificata in un singolo campione appartenente al lineage B.1.258. È associata ad un aumento dell’affinità per il recettore umano ACE2 ed è in grado di evadere l’attività neutralizzante di alcuni anticorpi monoclonali e di anticorpi presenti nel siero di una parte di individui guariti dall’infezione.
- S477N. Mutazione nel dominio di legame al recettore. Identificata in 8 campioni appartenenti al lineage B.1.160 e B.1.160.7. Questa mutazione sembra essere associata ad un aumento dell’affinità per il recettore umano ACE2 (Grid Based Pharmacophore Model) ed è in grado di evadere l’attività neutralizzante di alcuni anticorpi monoclonali.
Figure (cliccare per ingrandirle)
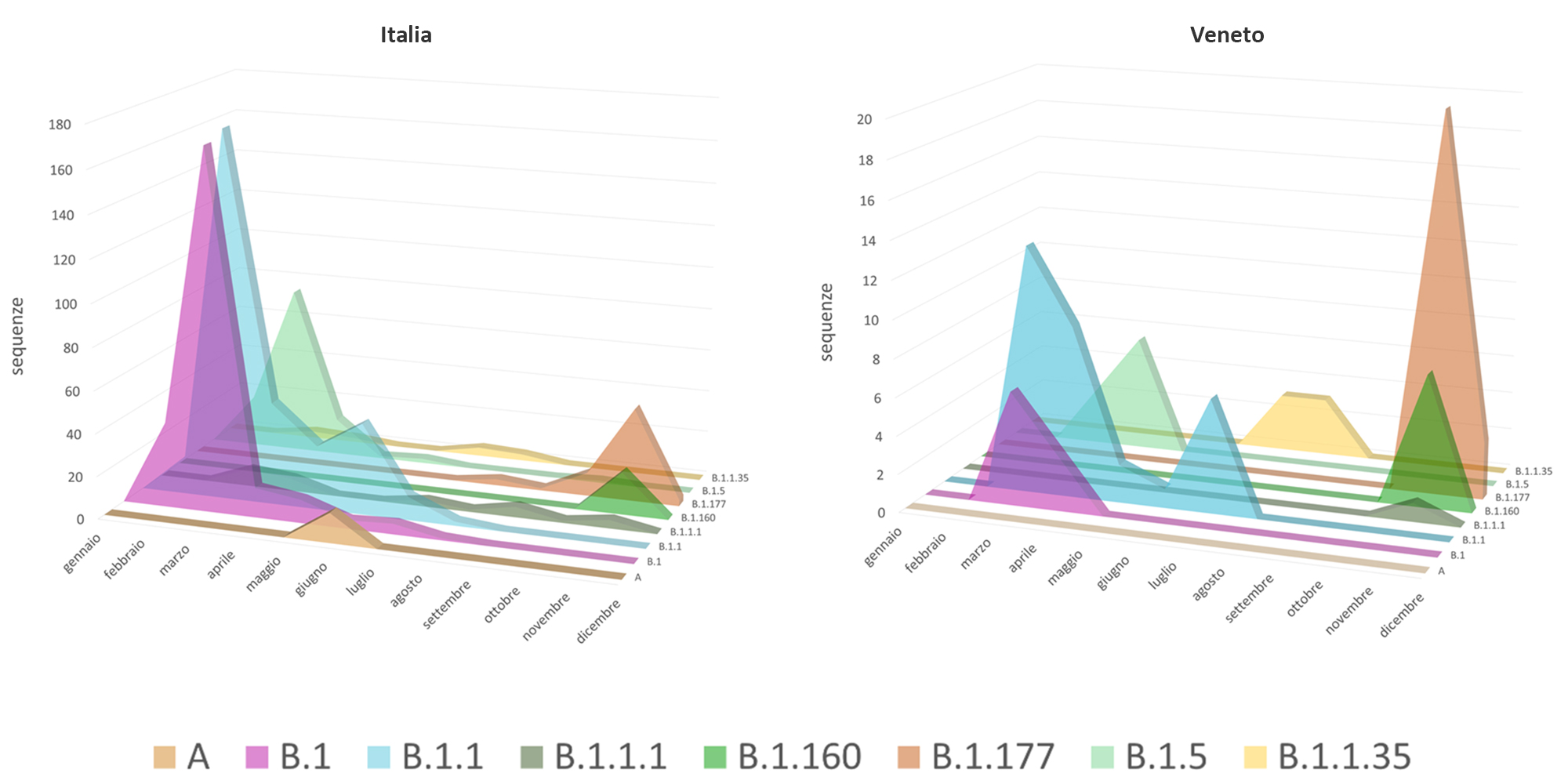
Figura 2. Distribuzione mensile dei principali lineage circolanti. A sinistra il dato relativo all’Italia mentre a destra quello relativo alla sola regione Veneto. Si può notare in entrambi i grafici che i genotipi B.1, B.1.5 e B.1.1 non sono più stati rilevati nella seconda metà dell’anno mentre sono invece diventati prevalenti i genotipi B.1.177 e B.1.160 (dati basati sulle sequenze italiane disponibili in GISAID in data 23/12/2020).
Referenze
- Bal A, Destras G, Gaymard A, Regue H, Semanas Q, d’Aubarde C, Billaud G, Laurent F, Gonzales C, Valette M, Lina B, Morfin F, Josset L. Screening of the H69 and V70 deletions in the SARS-CoV-2 spike protein with a RT-PCR diagnosis assay reveals low prevalence in Lyon, France. medRxiv 2020, 2020.11.10.20228528.
- Kemp, Steven A., Dami A. Collier, Rawlings Datir, Salma Gayed, Aminu Jahun, Myra Hosmillo, Isabella Atm Ferreira, et al. 2020. Neutralising Antibodies Drive Spike Mediated SARS-CoV-2 Evasion. medRxiv. https://www.medrxiv.org/content/10.1101/2020.12.05.20241927v1.full.
- Andrew Rambaut, Edward C. Holmes, Áine O’Toole, Verity Hill, John T. McCrone, Christopher Ruis, Louis du Plessis, and Oliver G. Pybus. 2020. A Dynamic Nomenclature Proposal for SARS-CoV-2 Lineages to Assist Genomic Epidemiology. Nature Microbiology 5 (11): 1403–7.
- Thomson EC, Rosen LE, Shepherd JG, Spreafico R. 2020. The Circulating SARS-CoV-2 Spike Variant N439K Maintains Fitness While Evading Antibody-Mediated Immunity. bioRxiv. https://www.biorxiv.org/content/10.1101/2020.11.04.355842v1.abstract.
- Zhuoming Liu, Laura A. VanBlargan, Paul W. Rothlauf, Louis-Marie Bloyet, Rita E. Chen, Spencer Stumpf, Haiyan Zhao, John M. Errico, Elitza S. Theel, Ali H. Ellebedy, Daved H. Fremont, Michael S. Diamond, Sean P. J. Whelan. Landscape analysis of escape variants identifies SARS-CoV-2 spike mutations that attenuate monoclonal and serum antibody neutralization. bioRxiv 2020.11.06.372037; doi: https://doi.org/10.1101/2020.11.06.372037
- Francesco Ortuso, Daniele Mercatelli, Pietro Hiram Guzzi, Federico Manuel Giorgi. Structural Genetics of circulating variants affecting the SARS-CoV-2 Spike / human ACE2 complex. bioRxiv 2020.09.09.289074; doi: https://doi.org/10.1101/2020.09.09.289074