L’interesse della comunità scientifica nei confronti dei tumori del cane è in crescita; non solo perché il cane è il migliore amico dell’uomo, ma anche perché costituisce un modello animale per lo studio comparato delle neoplasie nell’uomo.
Il sequenziamento del genoma canino e lo sviluppo delle recenti tecnologie NGS (Next Generation Sequencing) come RNA-seq oggi consentono ai ricercatori di studiare più specificamente i meccanismi molecolari di iniziazione e progressione delle neoplasie; inoltre, risultano utili per identificare potenziali biomarcatori prognostici e/o nuovi bersagli terapeutici. Studi di trascrittomica sono già stati condotti su alcune delle più importanti neoplasie canine, tra cui il linfoma e il mastocitoma. Poco si sa invece sulla biologia molecolare del melanoma orale, la neoplasia del cavo orale più comune nei cani.
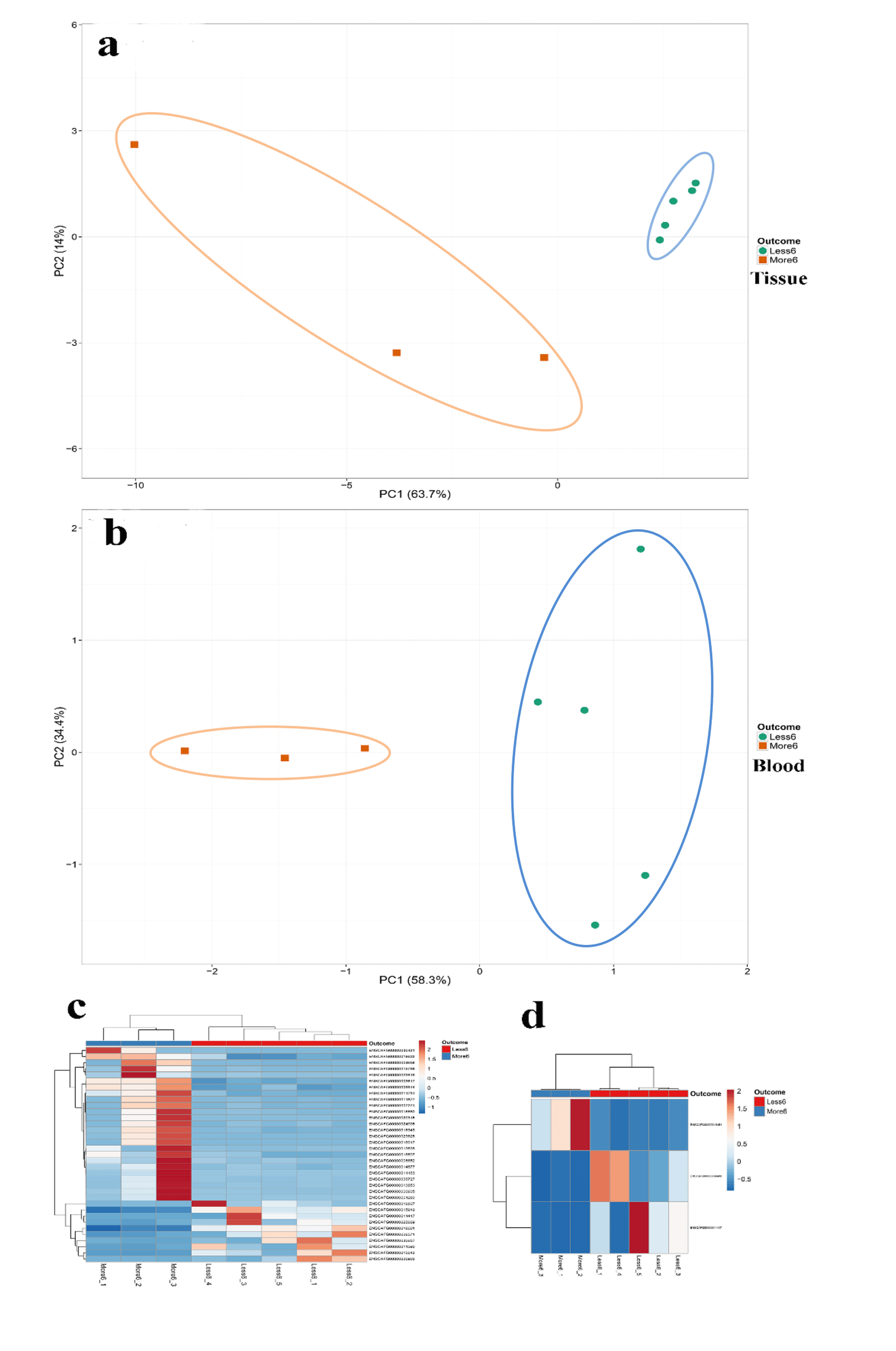
Una ricerca dell’IZSVe ha caratterizzato il trascrittoma del melanoma orale del cane (MOC) prima e dopo l’adozione di uno specifico approccio terapeutico. La ricerca ha pertanto evidenziato, particolarmente nel tessuto bioptico, un set di GDE che, se confermati in una ulteriore coorte di campioni, potrebbe essere utilizzato per predire il comportamento biologico del melanoma orale del cane. La figura rappresenta le analisi delle componenti principali (A,B) e le heatmap di clustering gerarchico (C,D) di campioni bioptici ed ematici ottenuti da cani affetti da MOC e stratificati in funzione dell’outcome della malattia.
Il melanoma orale è un tumore molto aggressivo, con un’alta propensione a creare metastasi al linfonodo locale e ai polmoni. Alcune razze canine sono più predisposte a sviluppare il melanoma: tra queste Scottish terrier, golden retriever, pastore di Beauce, bassotto, barbonicino, rottweiler, abrador retriever e schanuzer.
La chirurgia, la radioterapia e la chemioterapia non sembrano in grado di prolungare in maniera significativa la sopravvivenza dei cani con un melanoma orale in stadio avanzato; appare pertanto evidente che le conoscenze biologiche e molecolari su questa neoplasia necessitano di ulteriori approfondimenti.
La ricerca IZSVe
Una ricerca dell’Istituto Zooprofilattico Sperimentale delle Venezie (RC 11/13), finanziata dal Ministero della Salute, ha caratterizzato il trascrittoma del melanoma orale del cane prima e dopo l’adozione di uno specifico approccio terapeutico. Biopsie di tessuto buccale e sangue intero di cani affetti da melanoma orale sono state prima sottoposte ad indagini di RNA-seq; quindi i dati relativi al trascrittoma sono stati correlati con quelli del follow up clinico.
Sono stati raccolti 19 campioni di melanoma orale del cane, con stadiazione e follow up clinico completi. Al fine di ottenere la maggior omogeneità della coorte di campioni utilizzata, i campioni analizzati sono stati ridotti ad 8; questi comprendevano pazienti oncologici sottoposti a radioterapia e terapia adiuvante con temozolomide per i quali fosse noto il tempo di sopravvivenza e la causa della morte.
Risultati
Le analisi di RNA-seq su campione bioptico non hanno evidenziato geni differenzialmente espressi (GDE) prima e dopo la radioterapia; al contrario, nel sangue ne sono stati identificati 6. L’analisi delle componenti principali (PCA) ha distinto i campioni pre- e post-radioterapia, ma non è risultata particolarmente significativa.
La mancanza di differenze nel trascrittoma dei cani trattati con radioterapia suggerirebbe che la radioterapia non sia in grado di provocare modificazioni trascrizionali nel sito di irradiazione e nemmeno in un altro tessuto (sangue).
Risultati più interessanti si sono ottenuti quando si sono considerati l’outcome della malattia e la causa della morte. Adottando come criterio temporale il decesso entro 6 mesi, le analisi bioinformatiche ha distinto i campioni in due gruppi diversi: uno comprendente cani deceduti per melanoma entro 6 mesi, l’altro con cani ancora vivi a 6 mesi di follw-up o deceduti per cause diverse dal melanoma.
Queste analisi hanno consentito di individuare 35 e 3 GDE rispettivamente nel tessuto bioptico e nel sangue. Un unico gene, che codifica per la proteina ribosomiale L4 (RPL4), ha mostrato un comportamento simile (up-regulation) in entrambe le matrici.
La PCA ha distinto chiaramente i due set di campioni in entrambe le matrici, e la somma delle prime due componenti, pari a 77.7% per le biopsie e a 92.7% per il sangue, è risultata significativa. Questo dato è stato confermato anche dalle heatmaps di clustering gerarchico.
Conclusioni
La ricerca ha pertanto evidenziato, particolarmente nel tessuto bioptico, un set di GDE che, se confermati in una ulteriore coorte di campioni, potrebbe essere utilizzato per predire il comportamento biologico del melanoma orale del cane.
Particolare interesse potrebbe assumere il gene codificante per la proteina ribosomiale L4, che potrebbe rappresentare in prospettiva un biomarcatore misurabile in una matrice non invasiva come il sangue.




![Come proteggere il cane dalla leishmaniosi? [Video]](https://www.izsvenezie.it/wp-content/uploads/2024/05/come-proteggere-cane-leishmaniosi-video-500x383.jpg)


