Servizio di genotipizzazione di micoplasmi

Il Laboratorio di referenza WOAH per le micoplasmosi aviarie (M. gallisepticum, M. synoviae) ha messo a punto delle metodiche molecolari per genotipizzare i ceppi di Mycoplasma. Il servizio viene effettuato dai laboratori della SCT1 – Verona e della SCS5 – Ricerca e innovazione.
Cos’ è la genotipizzazione?
La genotipizzazione è un processo che permette di determinare differenze nel corredo genetico di un microorganismo.
La genotipizzazione consente di confrontare le similitudini e le differenze a livello genomico tra due o più microorganismi appartenenti alla stessa specie. L’analisi mette in relazione i microrganismi consentendo un raggruppamento tra ceppi geneticamente correlati. Ad ogni ceppo corrisponde un genotipo che lo caratterizza.
A cosa serve la genotipizzazione?
La genotipizzazione è molto utile in diversi ambiti. Ad esempio, conoscendo il genotipo di uno o più ceppi di microorganismi della stessa specie è possibile studiare gli aspetti epidemiologici di un’infezione. Infatti, è possibile stabilire le relazioni clonali tra ceppi, studiare popolazioni microbiche a livello di focolaio, a livello nazionale o addirittura globale, nonché ipotizzare o determinare la fonte di infezione o la cross-trasmissione di un microorganismo. Inoltre, tramite la genotipizzazione potrebbe essere possibile correlare determinati genotipi con determinate caratteristiche di virulenza oppure discriminare ceppi vaccinali da ceppi di campo.
Pertanto, avvalendosi della tecnica di genotipizzazione appropriata è possibile comprendere meglio l’epidemiologia del patogeno studiato e quindi porre in essere misure volte al contenimento della diffusione dei batteri patogeni, e quindi l’incidenza di nuovi focolai d’infezione.
Analisi di genotipizzazione disponibili
Genotipizzazione di M. gallisepticum
- Sequenziamento del gene mgc2 (GTS-mgc2)
- Multi Locus Sequence Typing (MLST) a 6 geni
Genotipizzazione di M. synoviae
- Sequenziamento del gene vlhA (GTS-vlhA)
- Multiple Locus Variable-Number Tandem Repeat Analysis
(MLVA) a 6 loci - Multi Locus Sequence Typing (MLST) a 7 geni
Per informazioni sul servizio
Elisabetta Stefani
SCT1 – Verona
Istituto Zooprofilattico Sperimentale delle Venezie
Via Bovolino, 1/C – 37060 Buttapietra (VR)
Tel. 045 500285 | estefani@izsvenezie.it
Ulteriori informazioni sul costo delle analisi, come raccogliere e consegnare i campioni, come effettuare i pagamenti e come consultare gli esiti degli accertamenti richiesti sono disponibili nelle pagine:
Dettagli sulle analisi disponibili
Mycoplasma gallisepticum | GTS-mgc2
Il genotipo del Mycoplasma gallisepticum viene identificato analizzando la sequenza nucleotidica ottenuta amplificando e sequenziando una porzione del gene codificante per la citoadesina mgc2. Campioni diagnostici (es. tamponi tracheali) e colture pure di M. gallisepticum possono essere processate tramite questo metodo.
Il genotipo (type) si ottiene confrontando la sequenza in esame con sequenze di riferimento secondo lo schema riportato in tabella e descritto qui: https://doi.org/10.3390/vaccines8040665. Per semplicità, ad ogni type viene assegnato un colore che corrisponde ad una sequenza di riferimento.
Il ceppo vaccinale 6/85 di M. gallisepticum viene identificato da questa metodica come type Orange. Fino ad oggi, nel territorio italiano non sono stati rilevati isolati di campo che vengono classificati come type Orange. Pertanto, sulla base delle evidenze epidemiologiche, attualmente è possibile utilizzare tale metodica per eseguire degli screening e discriminare il ceppo vaccinale dai ceppi di campo.
Invece, il ceppo vaccinale ts-11 di Mycoplasma gallisepticum viene identificato come type Pink, al quale appartengono anche alcuni ceppi di campo. In questo caso, quindi, non è possibile ottenere una discriminazione certa tra ceppo vaccinale ts-11 e ceppi di campo ed è necessario ricorrere alla metodica MLST a 6 geni di Mycoplasma gallisepticum per una più accurata discriminazione.
Per approfondire leggi l’articolo: Micoplasmosi aviarie, l’importanza della differenziazione molecolare di Mycoplasma gallisepticum nel controllo della malattia
Leggi l’articolo »| Ceppo | mgc2 type | GenBank Accession no. |
|---|---|---|
| TS-11 | PINK | JQ770175.1 |
| IZSVE/6344/3 | LIGHT-BLUE | KM107803 |
| 6/85 | ORANGE | JQ770178.1 |
| ATCC 15302 (Strain S6) | GREEN | KF874282.1 |
| c1- 2522/d1 | GREY | AY556297.1 |
| IZSVE/1464/2 | WHITE | KM107804 |
| IZSVE/AS958 | FUCSIA | KM107805 |
| IZSVE/2911/21 | BLACK | KM107806 |
| NCTC 10115 (Strain PG31) | VIOLET | AY556239.1 |
| IZSVE/AS24864 | BLUE | KM107808 |
| IZSVE/AS23252 | YELLOW | KM107807 |
| IZSVE/335/14 | PALE GRAY | – |
| IZSVE/1078/VR24/21 | ROSE PINK | – |
| IZSVE/5367/VR22/140 | LIGHT PINK | – |
| IZSVE/3405/VR23/6 | DARK PINK | – |
Mycoplasma gallisepticum | MLST 6 geni
La metodica MLST MG consente di assegnare un Sequence Type (ST) al campione di Mycoplasma gallisepticum sottoposto ad analisi. La metodica prevede l’amplificazione e il sequenziamento dei 6 geni (atpG2, dnaA, fusA, rpoB, ruvB, uvrA) costitutivamente espressi (housekeeping gene). Campioni diagnostici (es. tamponi tracheali) e colture pure di Mycoplasma gallisepticum possono essere processate tramite questo metodo.
Per l’assegnazione del ST viene utilizzato PubMLST, un database pubblico «open access» che attribuisce ad ogni allele sequenziato un numero. La combinazione dei 6 numeri ottenuti crea un profilo allelico che genotipizza il ceppo con un ST numerico. Attualmente questa metodica consente di discriminare i ceppi vaccinali 6/85 e ts-11 di Mycoplasma gallisepticum identificandoli con ST 14 e ST 49 rispettivamente. Inoltre l’alto potere discriminatorio della metodica risulta utile per la valutazione epidemiologica dei ceppi circolanti nel territorio italiano e nel mondo (https://doi.org/10.3390/vaccines8040665).
Per approfondire leggi l’articolo: Micoplasmosi aviarie, l’importanza della differenziazione molecolare di Mycoplasma gallisepticum nel controllo della malattia
Leggi l’articolo »Mycoplasma synoviae | GTS-vlhA
Il genotipo di Mycoplasma synoviae viene identificato analizzando una porzione del gene codificante per l’emoagglutinina vlhA che contiene una regione ricca di proline. Campioni diagnostici (es. tamponi tracheali) e colture pure di Mycoplasma synoviae possono essere processate tramite questo metodo.
Il genotipo (type) dei ceppi di campo si ottiene contando gli amminoacidi presenti nella regione PRR (Proline Rich Repeats), come descritto qui: https://doi.org/10.1016/j.vetmic.2008.10.011. In base al numero di amminoacidi rilevato viene assegnata una lettera dell’alfabeto, come indicato in tabella.
Sebbene il ceppo vaccinale MS-H di Mycoplasma synoviae appartenga al type C, per discriminarlo dai ceppi di campo che appartengono allo stesso genotipo, è necessario rilevare una perfetta omologia su tutto l’amplificato con la sequenza di referenza MS-H. Il ceppo viene quindi identificato come type C3-13.
Tuttavia, nel territorio italiano, in alcuni rari casi sono stati identificati ceppi di campo appartenenti al type C3-13. Pertanto, per una discriminazione più approfondita si può ricorrere all’analisi MLST a 7 geni o MLVA a 6 geni di Mycoplasma synoviae.
| vlhA type | Numero di amminoacidi del PRR |
|---|---|
| A | 38 |
| B | 45 |
| C | 32 |
| D | 23 |
| E | 19 |
| F | 36 |
| L | 35 |
| M | 41 |
| O | 31 |
| P | 27 |
| Q | 24 |
| R | 37 |
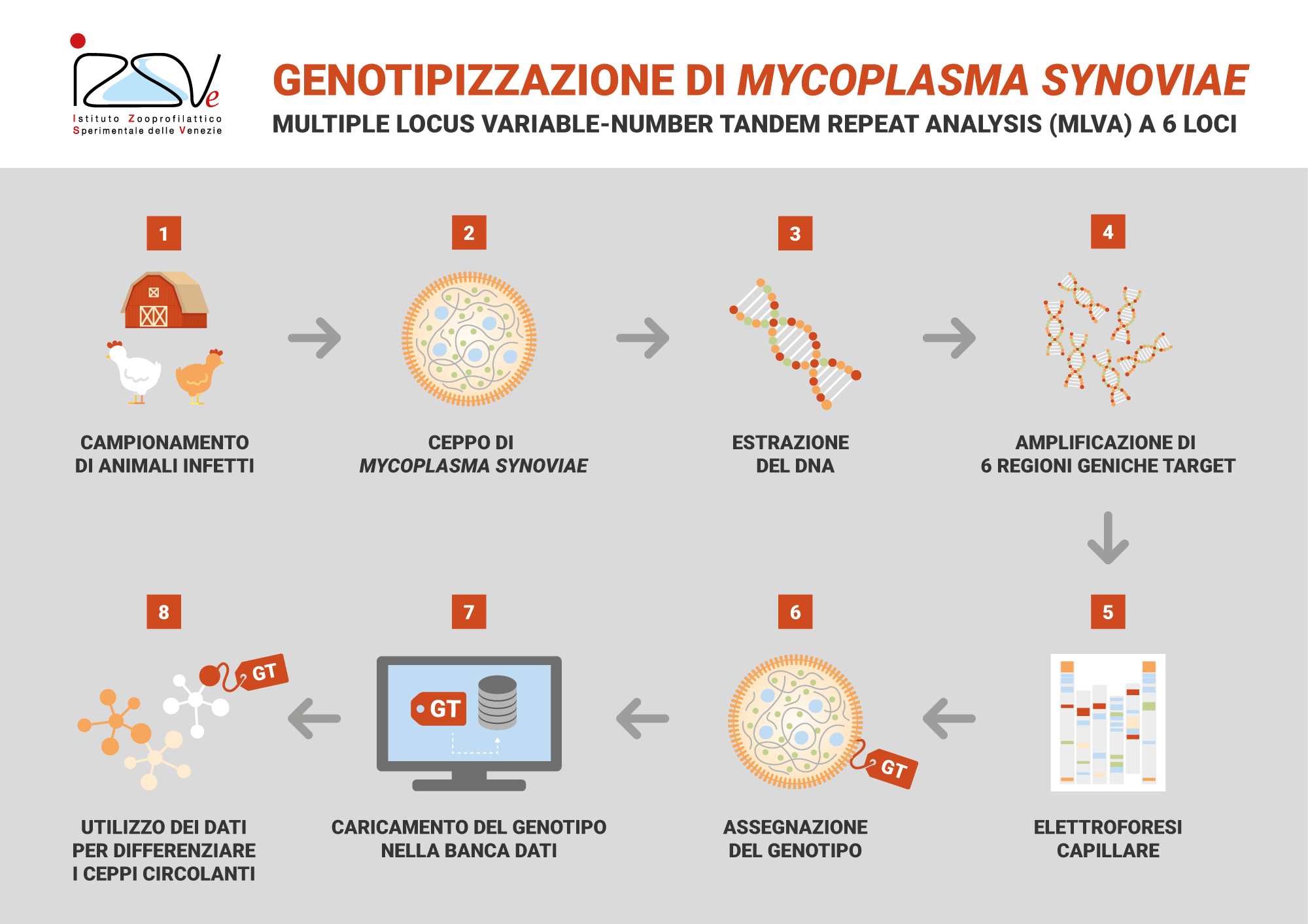
Mycoplasma synoviae | MLVA a 6 loci
La metodica prevede l’amplificazione di 6 regioni presenti nel genoma di M. synoviae. Ogni regione, detta locus, presenta ripetizioni di corte sequenze caratteristiche ripetute in tandem (tandem repeats, TR) che consentono la genotipizzazione del ceppo in esame. La metodica viene eseguita come di seguito descritto.
Campioni diagnostici
Possono essere sottoposti ad analisi MLVA sia campioni diagnostici (es. tamponi tracheali) che colture pure di Mycoplasma synoviae.
Estrazione e amplificazione del DNA
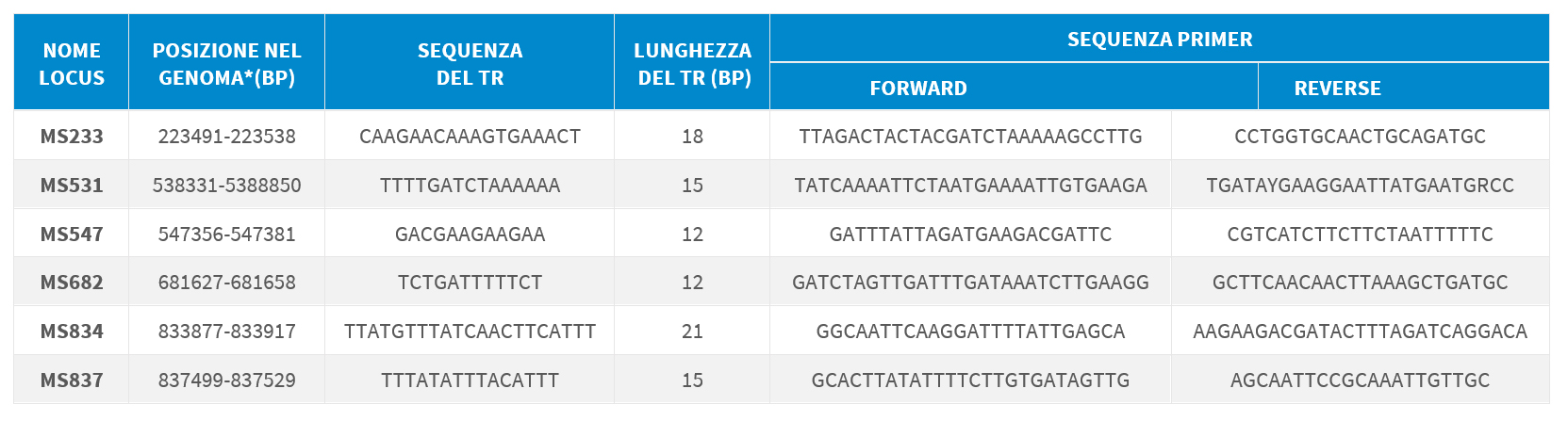
Il DNA genomico dei ceppi viene estratto utilizzando l’estrattore Maxwell 16 System e il kit Maxwell 16 Blood Purification System (Promega) oppure con il kit MagMax Pathogen RNA/DNA kit (Applied Biosystems) seguendo le istruzioni suggerite dal produttore. L’acido nucleico ottenuto viene amplificato nei 6 loci utilizzando i primer riportati in tabella. L’amplificazione viene eseguita con il kit Go Taq Polymerase (Promega). Il ciclo di amplificazione utilizzato prevede l’attivazione della Taq polimerasi a 95°C per 5’ seguita da 35 cicli a 95°C per 30”, 56°C per 30” e 72°C per 45”. Infine uno step di elongazione di 72°C per 5’ per tutti i loci tranne il MS531 in cui la temperatura di annealing è 58°C.
Elettroforesi capillare
Tutti gli amplificati ottenuti sono rilevati utilizzando l’elettroforesi capillare “Qiaexcel advanced System” della ditta Qiagen e il kit “Qiaexcel DNA Resolution kit” con il metodo OM800.
Le dimensioni ottenute sono comparate con il marcatore di peso molecolare 50-800 bp e con dei ceppi di riferimento specifici per ogni locus.
Interpretazione del risultato e assegnazione del genotipo
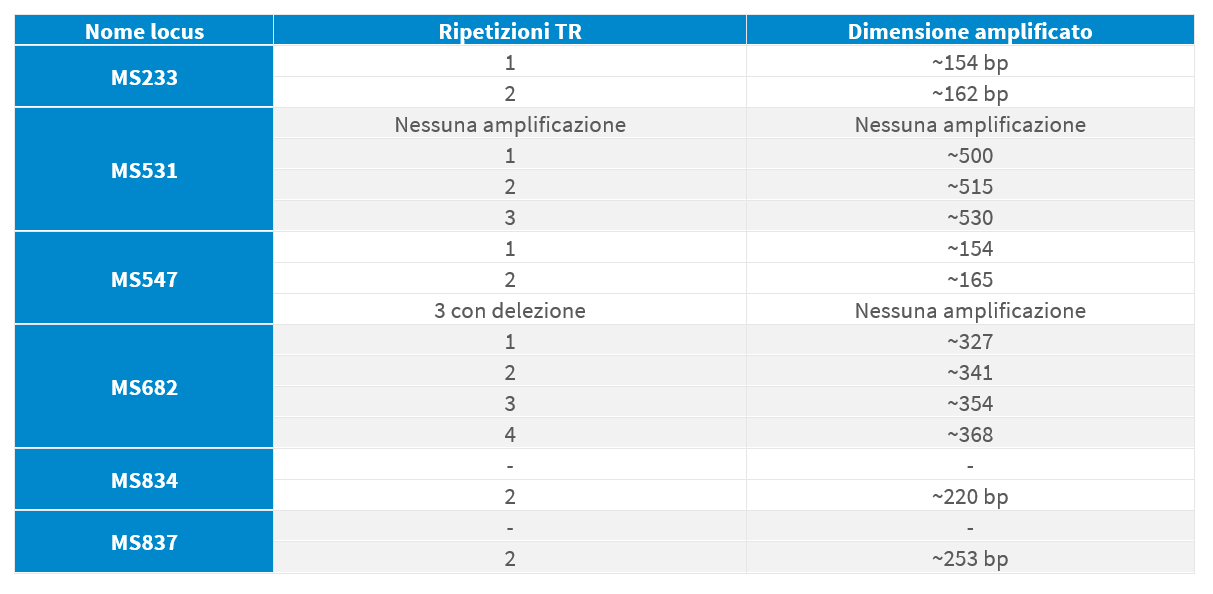
L’assegnazione del genotipo (GT) avviene in base al numero di TR ottenute in ogni locus amplificato. Dalla dimensione finale di ogni singolo amplificato si deduce il numero di TR presenti nel singolo locus. In base al numero di TR viene assegnato un numero ad ogni locus, oppure il valore 0 quando si registra assenza di amplificazione.
Il numero di TR finora riscontrati per ogni locus è riportato nella tabella 2. La combinazione dei numeri ottenuti per i 6 loci definisce univocamente il Genotipo (GT) del ceppo in esame; confrontando tale sequenza numerica con il database MLVA sarà possibile identificare il GT. I GT vengono identificati con numeri arabi (es., GT1, GT2, GT3, ecc.).
Database
Per questa metodica esiste un database utilizzabile per consultare, confrontare e depositare nuove sequenze (classificazione univoca dei ceppi).
Database IZSVe
* Utilizzare le frecce poste al lato dello grafico per visualizzare i diversi tipi di ceppo
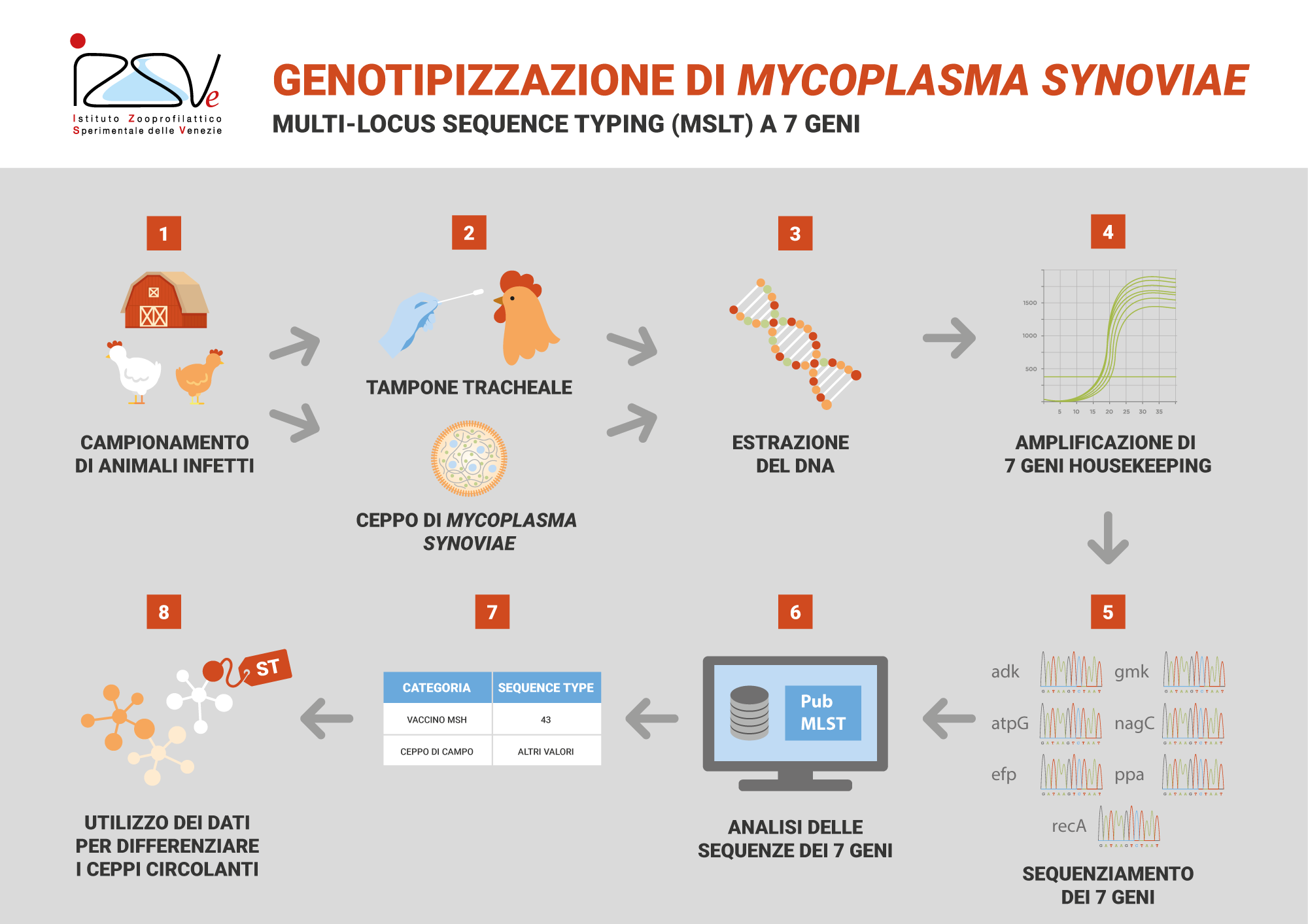
Mycoplasma synoviae | MLST 7 geni
La metodica MLST MS consente di assegnare un Sequence Type (ST) al campione di Mycoplasma synoviae sottoposto ad analisi. La metodica prevede l’amplificazione e il sequenziamento dei 7 geni (adk, atpG, efp, gmk, nagC, ppa e recA) costitutivamente espressi (housekeeping gene).
Campioni diagnostici (es. tamponi tracheali) e colture pure di Mycoplasma synoviae possono essere processate tramite questo metodo.
Per l’assegnazione del ST viene utilizzato PubMLST, un database pubblico «open access» che attribuisce ad ogni allele sequenziato un numero. La combinazione dei 7 numeri ottenuti crea un profilo allelico che genotipizza il ceppo con un ST numerico.
Attualmente questa metodica consente di discriminare il ceppo vaccinale MS-H di Mycoplasma synoviae identificandolo con ST 43. Inoltre l’alto potere discriminatorio della metodica risulta utile per la valutazione epidemiologica dei ceppi circolanti nel territorio italiano e nel mondo (https://doi.org/10.1637/11417-040516-reg).